•
Fosforylacja
(seryna, treonina)
•
Acetylacja
(lizyna)
•
Metylacja
(lizyna, arginina)
•
ADP-rybozylacja
•
Ubikwitynylacja
(lizyna)
•
Sumoilacja
(lizyna)
Modyfikacje histonów rdzeniowych
Modyfikacja
Modyfikowane reszty
aminokwasowe
Funkcja biologiczna
Acetylacja
K-ac
Aktywacja transkrypcji, replikacja
DNA, naprawa DNA, składanie
chromatyny
Metylacja
K-me1, K-me2, K-me3
R-me1, R-me2s, R-me2a
Aktywacja, elongacja i represja
transkrypcji
Fosforylacja
S-ph, T-ph, Y-ph
Aktywacja i represja transkrypcji,
naprawa DNA, mitoza, apoptoza
Ubikwitynacja
K-ub
Aktywacja transkrypcji, mejoza
Sumoilacja
K-su
Represja transkrypcji
ADP-rybozylacja
E-ar
Aktywacja transkrypcji (?)
Biotynylacja
K-bio
Regulacja ekspresji genów,
odpowiedź na uszkodzenia DNA
Krotonylacja
K-cr
Aktywacja transkrypcji (?)
Deiminacja
R → Cit
Regulacja transkrypcji
Ubikwitynylacja
Sumoilacja
Modyfikacja wszystkich 4 histonów rdzeniowych,
związana przede wszystkim z represją transkrypcji
C. David Allis, «Epigenetics» Cold Spring Harbor Laboratory Press
Cell, Vol 109, 801-806, June 2002 Review Histone Methylation: Dynamic or Static? Andrew J. Bannister, Robert Schneider, and Tony Kouzarides
Metylacji w histonach rdzeniowych podlegają lizyny i argininy
Metylotransferazy lizynowe
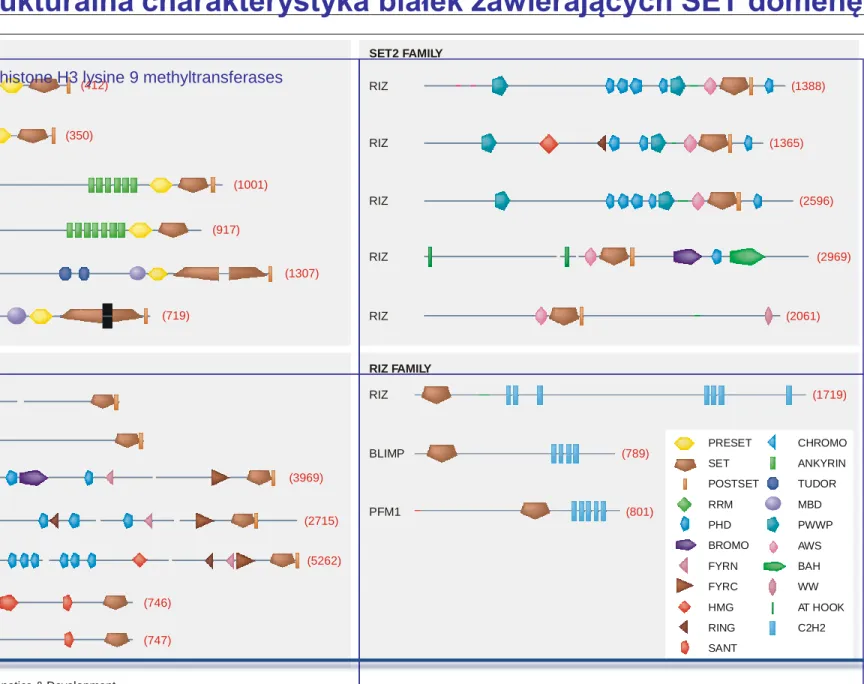
Figure 2 GLP ESET CLLL8 hSET1A RIZ BLIMP PFM1 RIZ RIZ RIZ RIZ RIZ hSET1B MLL MLL2 ALR EZH2 EZH1 SUV39H1 SUV39H2 G9A (1388) (1365) (2596) (2969) (2061) (1719) (789) (801) (3969) (2715) (5262) (746) (747) (412) (350) (1001) (917) (1307) (719) SET2 FAMILY SUV39 FAMILY RIZ FAMILY SET1 FAMILY PRESET SET POSTSET RRM PHD BROMO FYRN FYRC HMG RING SANT CHROMO ANKYRIN TUDOR MBD PWWP AWS BAH WW AT HOOK C2H2
Current Opinion in Genetics & Development
Strukturalna charakterystyka białek zawierających SET domenę
Roślinne białka z domeną SET
Ng et al.
2007
Lizynowe demetylazy histonowe
LSD1 (lysine-specific demethylase 1)
demetyluje mono- i di- metylowane lizyny
(H3K4me2/me1)
(H3K9me2/me1)
Rodzina Jumonji zawierająca domenę JmjC
(H3K9me3, H3K36me3)
Skład podjednostkowy kompleksów metylotransferaz H3K4
Current Opinion in Cell Biology 2008, 20:341–348
Mechanizmy rekrutacji metylotransferazy H3K4
3 (+1) główne polimerazy RNA
Eukaryota
•
Polimeraza mitochondrialna
polimeraza
produkty
Polimeraza I
geny rRNA (18S;
28S; 5,8S)
Polimeraza II
hn/mRNA,
większość snRNA
(U1, U2, U4, U5),
miRNA
Polimeraza III
małe RNA: tRNA,
snoRNA, 5S
Kluczowe znaczenie domeny karboksylowej (C-końcowej) RNA Pol II
•
“CTD” to domena C-końcowa największej
podjednostki RNA polimerazy II.
•
U człowieka liczy ponad 350 aminokwasów
złożonych z wielokrotnych powtórzeń
7-aminokwasowego motywu o sekwencji
konsensusowej: tyr-ser-pro-thr-ser-pro-ser.
•
CTD ma kluczowe znacznie dla
mechanizmów, które łączą zdarzenia
po-transkrypcyjne z transkrypcją. Na przykład,
praktycznie wszystkie kompleksy
zaangażowane w obróbkę RNA (RNA
processing) zawierają składniki, które wiążą
się do CTD.
Saunders et al. Nature Reviews Molecular Cell Biology 7, 557–567 (August 2006) | doi:10.1038/nrm1981
Domena CTD polimerazy II RNA koordynuje wydarzenia transkrypcyjne
Domena CTD zawiera powtarzającą się
sekwencję aminokwasową (YSPTSPS)
Hyperfosforylacja domeny CTD determinuje
nowy zestaw regulatorów przyłączających
się do pol II i zaznacza przejście od inicjacji
do elongacji transkrypcji.
Zatrzymanie w pobliżu promotora i
uwolnienie promotora; przejście do fazy
produktywnej transkrypcji – jest zależne od
fosforylacji CTD
Metylacja H3K4 i H3K36 w procesie transkrypcji
Zależności pomiędzy modyfikacjami histonowymi
H3K9-SUV39H1 – pierwsza zidentyfikowana metylotransferaza histonowa
Regulation of chromatin structure by site-specific histone methyltransferases
Rea S, Eisenhaber F, O'Carroll D, Strahl BD, Sun ZW, Schmid M, Opravil S, Mechtler K, Ponting CP,
Allis CD, Jenuwein T.
Cell, Vol 107, 323-337, November 2001
Loss of the Suv39h Histone Methyltransferases
Impairs Mammalian Heterochromatin and Genome
Stability
Antoine H.F.M. Peters 51, Dónal O'Carroll 5 61, Harry Scherthan 62, Karl
Mechtler1, Stephan Sauer1, Christian Schöfer3, Klara Weipoltshammer3, Michaela Pagani1, Monika Lachner1, Alexander Kohlmaier1, Susanne Opravil1, Michael Doyle 61, Maria Sibilia 61, and Thomas Jenuwein1
Konstytutywna heterochoromatyna zawiera histon H3 metylowany w lizynie 9
Mysia metylotransferaza Suv39h metyluje Lizynę 9
Histonu H3 w obszarach pericentromerowych
Myszy pozbawione Suv39h wykazują obniżoną żywotność,ich genom jest
niestabilny co prowadzi do zwiększonego ryzyka powstawania nowotworów
U myszy pozbawionych Suv39h następują zaburzenia oddziaływań
HP1
obszary heterochromatyny
wyciszone obszary euchromatyny
chromatyna obszarów
aktywnych transkrypcyjnie
H3K9me
H3K9me
Histon H3 metylowany w
lizynie 9 zlokalizowany
jest na nieaktywnym
chromosomie X
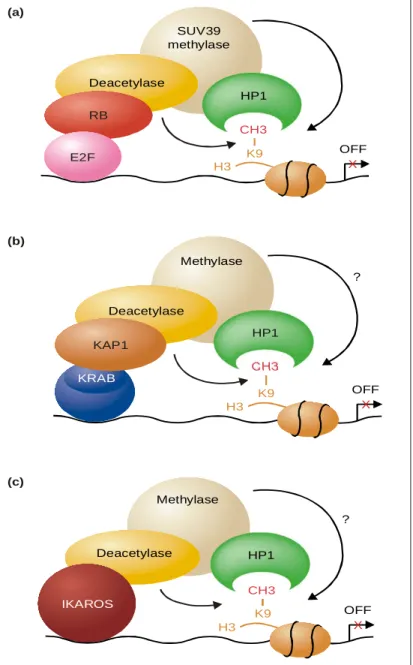
Rb targets histone H3 methylation and HP1 to promoters
SOREN J. NIELSEN*†, ROBERT SCHNEIDER*†, UTA-MARIA BAUER*, ANDREW J. BANNISTER*, ASHBY MORRISON‡, DONAL O'CARROLL§, RON FIRESTEIN, MICHAEL CLEARY,
THOMAS JENUWEIN§, RAFAEL E. HERRERA‡ & TONY KOUZARIDES* Nature 412, 561 - 565 (2001);
Metylacja lizyny 9 histonu H3 bierze udział w represji
transkrypcji
Białko Rb rekrutuje enzym deacetylazy histonowej HDAC i w ten sposób
reprymuje ekspresję genu
Represja genu cykliny E przez białko Rb (Retinoblastoma) jest
zależna od oddziaływań białka RB z białkami HP1 oraz Suv39H1.
Białko Rb kieruje metylacją histonu H3 i jest niezbędne dla
Figure 4 OFF H3 SUV39 methylase OFF H3 KRAB HP1 K9 CH3 ? ? Deacetylase RB E2F Methylase Methylase K9 CH3 Deacetylase Deacetylase HP1 KAP1 IKAROS HP1 K9 CH3 OFF H3 X X X (a) (b) (c)
Current Opinion in Genetics & Development
Przykłady represji transkrypcji
możliwe zaangażowanie
metylacji lizyny 9 histonu H3 w proces
represji
KAP1 – korepresor transkrypcji
kolokalizuje z białkiem HP1 w wielu
miejscach jądra interfazowego
IKAROS – represor transkrypcji,
rekrutuje deacetylazy histonowe,
kolokalizuje z białkiem HP1
Metylotransferazy lizynowe
Current Opinion in Cell Biology 2008, 20:341–348
Aktywacja transkrypcji
H3K4
H3K36
H3K79
H3K9
Represja transkrypcji
H3K9
H3K27
H4K20
Uzyskanie przeciwciał specyficznych w stosunku do metylowanej cytozyny w DNA,
a także poznanych potranslacyjnych modyfikacji histonów rdzeniowych
zrewolucjonizowało naszą wiedzę o chromatynie
Immunoprecypitacja
Oczyszczanie DNA Amplifikacja
Hybrydyzacja do mikromacierzy
Mapowanie rejonów w genomie
Immunoprecypitacja Oczyszczanie DNA Dołączenie sekwencji adaptorowych Tworzenie zbiorów Sekwencjonowanie
ChIP–chip
ChIP– Seq
ChIP – chromatin immunoprecipitation
Mapowanie rejonów w genomie
Rola modyfikacji potranslacyjnych histonów w regulacji
procesu transkrypcji
Modyfikacje histonowe wyznaczają funkcjonalne
elementy w genomie ssaków
Domeny białkowe specyficznie wiążące modyfikowane histony
Identyfikacja nowych białek rozpoznających określone
modyfikacje potranslacyjne białek histonowych
Joanna Wysocka, Methods 40 (2006) 339-343
Chromatografia powinowactwa do peptydu
Identyfikacja białek oddziałujących z metylowanymi lizynami histonu H3
Badanie roli metylacji lizyn histonu H3
Aktywacja transkrypcji
H3K4me3
Elongacja transkrypcji
H3K36me3
Wyciszenie transkrypcji
H3K9me3
H3K27me3
H4K20m3
SILAC – ang. stable isotope labeling by amino acids in cell culture
Cell 142, 967–980, September 17, 2010
Porównawcza
Spektrometria Mas
Cell 142, 967–980, September 17, 2010
Cell 142, September 17, 2010
Białka „odczytujące” wzór modyfikacji histonowych decydują
o efekcie biologicznym danej modyfikacji
Poszukiwanie partnerów białkowych dla modyfikowanych
potranslacyjnie histonów rdzeniowych
Utworzenie listy białek jądrowych specyficznie wiążących
modyfikowany potranslacyjnie histon H3 u Arabidopsis thaliana
Identyfikacja białek wiążących di-metylowany
(K4 lub K9) histon rdzeniowy H3 u roślin za pomocą techniki chromatografii
powinowactwa do peptydu (peptide pull-down assay).
.
Wykorzystywane peptydy
biotyn
a
H3 K9 me2
biotyn
a
H3 K4
me2
biotyn
a
H3
Materiał badawczy
Fot. Maciej Kotliński
Komórki Arabidopsis thaliana T87 ekotyp Columbia-0 (~8-dniowe).
F ot. Ma ci ej K otl ińs ki
Poszukiwanie unikatowych partnerów modyfikowanych peptydów po
rozdziale związanych białek w żelu
ic K4 H3 m
1
2
AL4
(At2g26210)
i
AL6
(At2g02470)
oraz ~
AL1
lub
AL7
AL2
(At3g11200)
i
AL7
(At1g14510)
oraz ~
AL1
lub
AL6
1
2
• Rodzina białek Alfin1-like
• Zawierają domenę palca cynkowego typu PHD
(zinc-finger plant homeodomain)
Wielkoskalowa analiza białek wytrąconych z eluatu,
które wiążą modyfikowane peptydy
•
Pominięto rozdział eluatów w żelu.
•
Po elucji, białka wytrącano na granicy faz metanol/chloroform.
•
Wytrącone białka trawiono trypsyną i poddawano analizie za pomocą metod
spektrometrii mas.
•
Interakcję białek z peptydem stwierdzano na podstawie porównania parametru
Mascot score zidentyfikowanych białek z poszczególnych eluatów
•
Metodę zastosowano do identyfikacji partnerów histonu H3 dimetylowanego na
Klasa białek H3 H3K9me2
H3 i H3K9me2
Histony 5 33 1
Białka budujące kompleksy polimeraz RNA zależnych
od DNA 0 8 11
Deacetylazy histonowe z rodziny HD2 0 0 3
ATP-azy remodelujące chromatynę 0 5 0
Białka z rodziny 14-3-3 0 5 0
Podjednostki Mediatora 0 1 0
Specyficzne czynniki transkrypcyjne 6 1 7
Białka z domeną WD-40 0 3 3
Metylotransferaza DNA (CMT3) 0 1 0
Czynniki splicingowe 0 2 1
Białka uczestniczące w składaniu chromatyny 1 1 1
Topoizomerazy DNA 0 0 2
Białka chromatynowe związane z kompleksem
Polycomb 1 0 0
Białka wiążące DNA 1 1 3
Represory transkrypcji 0 1 0
Czynniki translacyjne 0 6 1
Białka budujące rybosomy 5 43 21
Małe jąderkowe nukleoproteiny 0 3 1
Białka wiążące RNA 2 1 4
Białka budujące cytoszkielet 0 4 18
Kinazy białkowe 1 4 0
Białka z domeną F-box 0 5 0
Inne 9 115 19
Nieznane 1 18 2